Các kiểu gen của Cirovirus và tác nhân đồng nhiễm gây bệnh đường hô hấp (PRDC) trên heo ở các tỉnh miền Nam Việt Nam
Porcine Cirovirus type 2 (PCV2) là một loại vi-rút nhỏ, không có vỏ bọc ngoài, bộ gien DNA sợi đơn thuộc họ Circoviridae. Hạt vi-rút có dạng đối xứng hình tứ diện, với đường kính khoảng 17 nm và chứa một bộ gien tròn với khoảng 1767-1768 nucleotide (nt) [10, 37]. Trong số mười một khung đọc mở (ORF) dự đoán, xếp chồng chéo và lồng vào nhau, sáu ORF (ORF1–6) đã được xác định là mã hóa các protein có liên quan đến sự nhân lên, cấu trúc, sinh bệnh, sự tồn tại của vi-rút trong tế bào và tương tác miễn dịch của vi-rút với vật chủ [13, 20, 30]. ORF2 mã hóa protein capsid của vi-rút, protein cấu trúc duy nhất của PCV2, chứa các yếu tố kháng nguyên quan trọng liên quan đến sự trung hòa vi rút [3, 29]. Do đó, gien ORF2 và protein Cap được mã hóa là các mục tiêu chính để phân tích sự đa dạng di truyền, phát sinh loài, dịch tễ học và cơ chế né tránh miễn dịch của PCV2 [34, 39].
PCV2 gây ra các rối loạn lâm sàng khác nhau và được gọi là bệnh liên quan đến Circovirus ở heo (PCVADs). PCVAD bao gồm hội chứng còi cọc trên heo sau cai sữa (PMWS), bệnh hô hấp phức hợp ở heo (PRDC), hội chứng viêm da và thận ở heo (PDNS), rối loạn sinh sản, tăng sinh và hoại tử viêm phổi (PNP), run bẩm sinh loại II (CT), và viêm ruột [32]. Một trong những lý do tại sao PCV2 đóng vai trò quan trọng trong các hội chứng khác nhau được đề cập ở trên là vì PCV2 tấn công các tế bào miễn dịch, dẫn đến ức chế miễn dịch và do đó mở đường cho các vi khuẩn và vi rút cơ hội khác gây bệnh [20, 23, 24]. Do đó, nhiễm PCV2 kết hợp với các mầm bệnh khác thường được quan sát thấy ở các trang trại heo, chẳng hạn như kết hợp với vi-rút gây Hội chứng rối loạn sinh sản và hô hấp trên heo (PRRSV) (42–85,4%) [7, 24], Mycoplasma hyopneumoniae (MH) (35,5%) [26], Haemophilus parasuis (HP) (32,3%) [17], và vi rút Dịch tả heo (CSFV – Classical Swine Fever Virus) [4]. Hiện tại, PCV2 được coi là một trong những loại vi rút quan trọng nhất lây nhiễm cho heo ở một số quốc gia, bao gồm cả Việt Nam, và bệnh liên quan đến Circovirus trên heo (PCVADs) có thể gây ra thiệt hại kinh tế đáng kể cho ngành chăn nuôi heo toàn cầu.
CSFV là mầm bệnh đã được kiểm soát hoặc bị loại bỏ ở nhiều quốc gia, nhưng vi-rút này vẫn hiện diện phổ biến ở Việt Nam, tham gia và làm phức tạp thêm các trường hợp PRDC. Ngoài ra, sự kết hợp của PCV2 và PRRSV đóng một vai trò chủ yếu trong những ca bệnh PRDC. Hiện tại, ở Việt Nam đã có nhiều loại vắc xin phòng những mầm bệnh này, đơn giá hoặc đa giá, nhưng PRDC vẫn là mối quan tâm lớn trong quá trình chăn nuôi heo. Mặc dù việc sử dụng vắc xin rộng rãi rõ ràng sẽ thuận lợi cho việc kiểm soát dịch bệnh, nhưng áp lực được tạo ra bởi việc tiêm chủng tại các trang trại heo, cùng với chọn lọc tự nhiên lâu dài, có thể dẫn đến sự biến đổi gien và sự thất bại của vắc xin, như đối với PCV2 đã được báo cáo trước đây [25, 33, 43]. PCV2 có tỷ lệ thay đổi nucleotide cao so với các vi-rút DNA khác. Tỷ lệ đột biến ước tính cho PCV2 được xác định là là 1,2 × 10-3 mỗi năm [8].
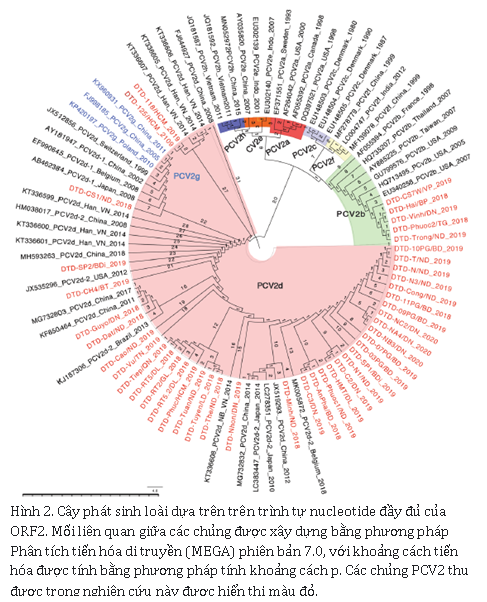
Lúc đầu, PCV2a là kiểu gien phổ biến nhất ở các quốc gia khác nhau [28], tiếp theo là kiểu gien PCV2b sau năm 2004 [16, 22], và sau đó đã chuyển sang PCV2d từ khoảng năm 2012 trở đi [40]. Ở Việt Nam, nghiên cứu hồi cứu chỉ ra rằng, PCV2 xuất hiện trước năm 2000 [40], và PCV2b chiếm ưu thế cho đến năm 2012 [41]. Sau đó, PCV2d được ghi nhận vào năm 2010 [42] và chiếm ưu thế sau năm 2014 [12]. Kết quả phân tích gien ORF2 của các chủng PCV2 cho thấy, hầu hết các trường hợp heo nhiễm PCV2 tại các trang trại ở Việt Nam là do kiểu gien PCV2d, tiếp theo là PCV2b. PCV2d có thể được chia thành hai loại phụ: PCV2d-1 và PCV2d-2 [42]. PCV2d-2 phổ biến hơn PCV2d-1 [41, 42], điều này chỉ ra rằng, những thay đổi di truyền và xu hướng tiến hóa của PCV2 ở Việt Nam cũng tương tự như ở các các quốc gia khác, chẳng hạn như Trung Quốc, Hoa Kỳ và Hàn Quốc.
Sự đa dạng quan sát được ở PCV2 trên toàn thế giới, cùng với sự thay đổi kiểu gien từ PCV2a sang PCV2b từ năm 2004, và xa hơn nữa là PCV2d từ năm 2012, chủ yếu xảy ra ở heo thương phẩm, chứng minh sự tiến hóa liên tục của vi-rút này trong các đàn đã được gây miễn dịch. Hiện tượng này cũng cho thấy rõ khả năng xuất hiện của một kiểu gien PCV2 ưu thế mới, đặc biệt là sau khi có vắc-xin mới được giới thiệu vào thị trường.
Tài liệu tham khảo
1. Abhijit K, Barate H-YL, Jeong H-W, Truong LQ, Joo H-G, Hahn T-W (2012). An improved multiplex PCR for diagnosis and differentiationof Mycoplasma hyopneumoniae and Mycoplasmahyorhinis. Korean J Vet Res 52:39–43.
2. Allan GM, McNeilly F, Kennedy S, Daft B, Clarke EG, Ellis JA, Haines DM, Meehan BM, Adair BM (1998). Isolation of porcinecircovirus-like viruses from pigs with a wasting disease in theUSA and Europe. J Vet Diagn Invest 10:3–10.
3. Blanchard P, Mahe D, Cariolet R, Keranflec’h A, Baudouard MA, Cordioli P, Albina E, Jestin A (2003). Protection of swine againstpost-weaning multisystemic wasting syndrome (PMWS) by porcinecircovirus type 2 (PCV2) proteins. Vaccine 21:4565–4575.
4. Chen N, Huang Y, Ye M, Li S, Xiao Y, Cui B, Zhu J (2019). Co-infection status of classical swine fever virus (CSFV), porcinereproductive and respiratory syndrome virus (PRRSV) andporcine circoviruses (PCV2 and PCV3) in eight regions of Chinafrom 2016 to 2018. Infect Genet Evol 68:127–135.
5. Cheung AK, Lager KM, Kohutyuk OI, Vincent AL, Henry SC, Baker RB, Rowland RR, Dunham AG (2007). Detection of twoporcine circovirus type 2 genotypic groups in United States swineherds. Arch Virol 152:1035–1044.
6. Do DT, Park C, Choi K, Jeong J, Nguyen TT, Le DT, Vo KM, Chae C (2016). Nucleotide sequence analysis of Vietnamese highlypathogenic porcine reproductive and respiratory syndrome virusfrom 2013 to 2014 based on the NSP2 and ORF5 coding regions.Arch Virol 161:669–675.
7. Dorr PM, Baker RB, Almond GW, Wayne SR, Gebreyes WA (2007). Epidemiologic assessment of porcine circovirus type 2coinfection with other pathogens in swine. J Am Vet Med Assoc230:244–250.
8. Firth C, Charleston MA, Duffy S, Shapiro B, Holmes EC (2009). Insights into the evolutionary history of an emerging livestockpathogen: porcine circovirus 2. J Virol 83:12813–12821.
9. Franzo G, Segales J (2018) Porcine circovirus 2 (PCV-2) genotype update and proposal of a new genotyping methodology. PLoSONE 13:e0208585.
10. Hamel AL, Lin LL, Nayar GP (1998). Nucleotide sequence of porcine circovirus associated with postweaning multisystemicwasting syndrome in pigs. J Virol 72:5262–5267.
11. Huang LP, Lu YH, Wei YW, Guo LJ, Liu CM (2011). Identification of one critical amino acid that determines a conformationalneutralizing epitope in the capsid protein of porcine circovirustype 2. BMC Microbiol 11:188.
12. Huynh TM, Nguyen BH, Nguyen VG, Dang HA, Mai TN, Tran TH, Ngo MH, Le VT, Vu TN, Ta TK, Vo VH, Kim HK, ParkBK (2014). Phylogenetic and phylogeographic analyses of porcinecircovirus type 2 among pig farms in Vietnam. Transbound EmergDis 61:e25-34.
13. Juhan NM, LeRoith T, Opriessnig T, Meng XJ (2010) The open reading frame 3 (ORF3) of porcine circovirus type 2 (PCV2) isdispensable for virus infection but evidence of reduced pathogenicityis limited in pigs infected by an ORF3-null PCV2 mutant.Virus Res 147:60–66.
14. Kedkovid R, Woonwong Y, Arunorat J, Sirisereewan C, Sangpratum N, Lumyai M, Kesdangsakonwut S, Teankum K, JittimaneeS, Thanawongnuwech R (2018). Porcine circovirus type 3 (PCV3)infection in grower pigs from a Thai farm suffering from porcinerespiratory disease complex (PRDC). Vet Microbiol 215:71–76.
15. Khayat R, Brunn N, Speir JA, Hardham JM, Ankenbauer RG, Schneemann A, Johnson JE (2011). The 2.3-angstrom structureof porcine circovirus 2. J Virol 85:7856–7862.
16. Kim HK, Luo Y, Moon HJ, Park SJ, Keum HO, Rho S, ParkBK (2009). Phylogenetic and recombination analysis of genomicsequences of PCV2 isolated in Korea. Virus Genes 39:352–358
17. Kim J, Chung HK, Jung T, Cho WS, Choi C, Chae C (2002). Postweaning multisystemic wasting syndrome of pigs in Korea:prevalence, microscopic lesions and coexisting microorganisms.J Vet Med Sci 64:57–62.
18. Lekcharoensuk P, Morozov I, Paul PS, Thangthumniyom N, Wajjawalku W, Meng XJ (2004). Epitope mapping of the major capsidprotein of type 2 porcine circovirus (PCV2) by using chimericPCV1 and PCV2. J Virol 78:8135–8145.
19. Lv N, Zhu L, Li W, Li Z, Qian Q, Zhang T, Liu L, Hong J, Zheng X, Wang Y, Zhang Y, Chai J (2020). Molecular epidemiology andgenetic variation analyses of porcine circovirus type 2 isolatedfrom Yunnan Province in China from 2016–2019. BMC Vet Res 16:96.
20. Meng XJ (2013). Porcine circovirus type 2 (PCV2): pathogenesisand interaction with the immune system. Annu Rev Anim Biosci1:43–64.
21. Oliveira S, Galina L, Pijoan C (2001). Development of a PCR test to diagnose Haemophilus parasuis infections. J Vet Diagn Invest13:495–501.
22. Olvera A, Sibila M, Calsamiglia M, Segales J, Domingo M (2004). Comparison of porcine circovirus type 2 load in serum quantifiedby a real time PCR in postweaning multisystemic wasting syndromeand porcine dermatitis and nephropathy syndrome naturallyaffected pigs. J Virol Methods 117:75–80.
23. Opriessnig T, Gimenez-Lirola LG, Halbur PG (2011). Polymicrobialrespiratory disease in pigs. Anim Health Res Rev 12:133–148.
24. Opriessnig T, Halbur PG (2012). Concurrent infections are important for expression of porcine circovirus associated disease. VirusRes 164:20–32.
25. Opriessnig T, Xiao CT, Gerber PF, Halbur PG (2013). Emergence of a novel mutant PCV2b variant associated with clinical PCVADin two vaccinated pig farms in the U.S. concurrently infected withPPV2. Vet Microbiol 163:177–183.
26. Pallares FJ, Halbur PG, Opriessnig T, Sorden SD, Villar D, Janke BH, Yaeger MJ, Larson DJ, Schwartz KJ, Yoon KJ, Hoffman LJ(2002). Porcine circovirus type 2 (PCV-2) coinfections in US fieldcases of postweaning multisystemic wasting syndrome (PMWS).J Vet Diagn Invest 14:515–519
27. Paton DJ, McGoldrick A, Bensaude E, Belak S, Mittelholzer C, Koenen F, Vanderhallen H, Greiser-Wilke I, Scheibner H, StadejekT, Hofmann M, Thuer B (2000). Classical swine fever virus:a second ring test to evaluate RT-PCR detection methods. Vet Microbiol 77:71–81.
28. Patterson AR, Opriessnig T (2010). Epidemiology and horizontal transmission of porcine circovirus type 2 (PCV2). Anim HealthRes Rev 11:217–234.
29. Pogranichnyy RM, Yoon KJ, Harms PA, Swenson SL, Zimmerman JJ, Sorden SD (2000). Characterization of immune responseof young pigs to porcine circovirus type 2 infection. Viral Immunol13:143–153.
30. Ren L, Chen X, Ouyang H (2016). Interactions of porcine circovirus 2 with its hosts. Virus Genes 52:437–444.
31. Saha D, Lefebvre DJ, Ooms K, Huang L, Delputte PL, Van Doorsselaere J, Nauwynck HJ (2012). Single amino acid mutations in thecapsid switch the neutralization phenotype of porcine circovirus2. J Gen Virol 93:1548–1555.
32. Segales J (2012). Porcine circovirus type 2 (PCV2) infections: clinical signs, pathology and laboratory diagnosis. Virus Res164:10–19.
33. Seo HW, Han K, Park C, Chae C (2014). Clinical, virological, immunological and pathological evaluation of four porcine circovirustype 2 vaccines. Vet J 200:65–70.
34. Ssemadaali MA, Ilha M, Ramamoorthy S (2015). Genetic diversity of porcine circovirus type 2 and implications for detection andcontrol. Res Vet Sci 103:179–186.
35. Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S (2011). MEGA5: molecular evolutionary genetics analysis usingmaximum likelihood, evolutionary distance, and maximum parsimonymethods. Mol Biol Evol 28:2731–2739.
36. Thompson JD, Higgins DG, Gibson TJ (1994). CLUSTAL W:improving the sensitivity of progressive multiple sequencealignment through sequence weighting, position-specific gap penaltiesand weight matrix choice. Nucleic Acids Res 22:4673–4680.
37. Tischer I, Gelderblom H, Vettermann W, Koch MA (1982). A very small porcine virus with circular single-stranded DNA. Nature295:64–66.
38. Trible BR, Kerrigan M, Crossland N, Potter M, Faaberg K, Hesse R, Rowland RR (2011). Antibody recognition of porcine circovirustype 2 capsid protein epitopes after vaccination, infection, anddisease. Clin Vaccine Immunol 18:749–757.
39. Trible BR, Rowland RR (2012). Genetic variation of porcine circovirus type 2 (PCV2) and its relevance to vaccination, pathogenesisand diagnosis. Virus Res 164:68–77.
40. Xiao CT, Halbur PG, Opriessnig T (2012). Complete genome sequence of a novel porcine circovirus type 2b variant present incases of vaccine failures in the United States. J Virol 86:12469.
41. Xiao CT, Halbur PG, Opriessnig T (2015). Global molecular genetic analysis of porcine circovirus type 2 (PCV2) sequencesconfirms the presence of four main PCV2 genotypes and revealsa rapid increase of PCV2d. J Gen Virol 96:1830–1841.
42. Xiao CT, Harmon KM, Halbur PG, Opriessnig T (2016). PCV2d-2 is the predominant type of PCV2 DNA in pig samples collectedin the U.S. during 2014–2016. Vet Microbiol 197:72–77.
43. Xu PL, Zhao Y, Zheng HH, Tian RB, Han HY, Chen HY, Zheng LL (2019). Analysis of genetic variation of porcine circovirustype 2 within pig populations in central China. Arch Virol164:1445–1451.
44. Yao J, Qin Y, Zeng Y, Ouyang K, Chen Y, Huang W, Wei Z (2019). Genetic analysis of porcine circovirus type 2 (PCV2) strainsbetween 2002 and 2016 reveals PCV2 mutant predominating in .porcine population in Guangxi, China. BMC Vet Res 15:118
45. Zhan Y, Wang N, Zhu Z, Wang Z, Wang A, Deng Z, Yang Y (2016). In silico analyses of antigenicity and surface structurevariation of an emerging porcine circovirus genotype 2b mutant,prevalent in southern China from 2013 to 2015. J Gen Virol97:922–933.
46. Zheng G, Lu Q, Wang F, Xing G, Feng H, Jin Q, Guo Z, Teng M, Hao H, Li D, Wei X, Zhang Y, Deng R, Zhang G (2020). Phylogeneticanalysis of porcine circovirus type 2 (PCV2) between 2015and 2018 in Henan Province, China. BMC Vet Res 16:6.
Nguồn: Sưu tầm
